3次元点群データ(ポイントクラウド)の解析¶
このチュートリアルでは、3次元点群データに対するパーシステントホモロジー解析の基本的な手法について解説します。
取り上げる主なトピックは以下の4点です:
- 3次元点群データからパーシステンス図(Persistence Diagram)を計算する
- パーシステンス図を可視化する
- 各ペアの birth/death time を抽出する
- 各ペアから元の構造を特定する逆解析
これらの内容は、パーシステントホモロジー解析において広く利用される基本的なステップです。まずはこのチュートリアルを通じて、解析の流れと考え方を身につけましょう。
チュートリアルの末尾には、以下のような発展的な内容についての導入も含まれています:
- 頂点の命名
- Optimal 1-cycle
- PHTrees
- 初期半径
- PyVista の BackgroundPlotter
これらのトピックは、より高度な解析や可視化を行いたい場合に役立ちます。必要に応じてそれぞれの内容を学び活用してみてください。
点群データの読み込み¶
解析対象のデータは、pointcloud.txt というファイルに保存されています。
このファイルを用いて、点群データのパーシステンス図を計算してみましょう。
まずは、解析に必要な Python ライブラリをインポートします。
import homcloud.interface as hc # HomCloudのインターフェース
import numpy as np # NumPy (数値配列)
import matplotlib.pyplot as plt # Matplotlib (PDの可視化用)
import pyvista as pv # PyVista (3D可視化)
3次元可視化には PyVista を利用し、そのバックエンドとして trame を利用します。 詳細については、PyVista の公式ドキュメントをご参照ください。
なお、環境によっては trame が正しく動作しない場合があります。その際は、バックエンドを 'trame' から 'static' に変更してみてください。 この変更をすると利用可能な機能が減りますが、可視化が安定します。
pv.set_jupyter_backend('trame')
<MagicMock name='mock()' id='134036025104992'>
データの読み込みには、NumPy の np.loadtxt 関数を使用します。
pointcloud = np.loadtxt("pointcloud.txt")
読み込んだデータについて調べます。
pointcloud.shape
(1000, 3)
np.min(pointcloud, axis=0), np.max(pointcloud, axis=0)
(array([0.00026274, 0.00100671, 0.00020878]), array([0.99997803, 0.9993682 , 0.99825316]))
読み込んだデータは、形状が $1000 \times 3$ の配列です。これは、3次元空間上の点が 1000 個あることを意味します。
各点の X, Y, Z 座標の最小値と最大値を確認すると、データはおおよそ $[0, 1] \times [0, 1] \times [0, 1]$ の立方体領域内に分布していることがわかります。
それでは、この点群データを3次元で可視化してみましょう。
plotter = pv.Plotter()
plotter.add_mesh(pv.PointSet(pointcloud))
plotter.show()
<MagicMock name='mock()' id='134035003131328'>
パーシステンス図の計算¶
データからパーシステンス図を計算します。
この処理にはhc.PDList.from_alpha_filtrationメソッドを使用します。
引数 save_boundary_map=True を指定することで、後のステップで stable volume を計算するために必要な情報も保存されます。
hc.PDList.from_alpha_filtration(pointcloud,
save_to="pointcloud.pdgm",
save_boundary_map=True)
PDList(path=pointcloud.pdgm)
すると pointcloud.pdgm というファイルが生成されます。これが
パーシステンス図の情報を収めたファイルです。
pdlist = hc.PDList("pointcloud.pdgm")
このオブジェクトは、0次元から2次元までのすべての次元に対応するパーシステンス図を保持しています。
すべての次元の情報をまとめて扱うため、このクラスは PDList という名前になっています。
ここでは、1次元のパーシステンス図だけを取り出してみましょう。
pd1 = pdlist.dth_diagram(1)
このメソッドの返り値は、PD クラスのインスタンスです。
ここでは、ヒストグラムを作成し、それをもとにパーシステンス図をプロットしてみましょう。
ヒストグラムの構築には histogram メソッドを、プロットには plot メソッドを使用します。
pd1.histogram().plot()
現在の色使いは、あまり視認性が高いとは言えません。 パーシステンス図は、ログスケール(対数スケール)で表示すると、より見やすくなることが多いため、ここではログスケールを適用してみましょう。
pd1.histogram().plot(colorbar={"type": "log"})
基本的に、パーシステンス図における点は、対角線から離れているほど「意味のある」リング構造に対応しています。
また、Y軸の値が大きいほど、より大きなスケールのリング構造を表していると解釈できます。
たとえば、点 $(0.0025, 0.008)$ の付近にあるものは、最も明確で特徴的なリング構造に対応していると考えられます。
ここで注意すべき点として、パーシステンス図の X 軸・Y 軸は、各点に置かれる球の「半径」と対応していると、一般的な教科書では説明されています。
しかし、HomCloud では、標準では「半径の2乗」が使用されています。
つまり、
- $ \sqrt{0.0025} = 0.05 $
- $ \sqrt{0.008} \approx 0.089 $
が実際の半径になります。
これは主に内部アルゴリズムの都合によるものですが、点に重みを付ける場合には、2乗の値を使う方が自然に見えるという理由もあります。
なお、この挙動を変更したい場合は、hc.PDList.from_alpha_filtration を使用してパーシステンス図を計算する際に、引数 squared=False を指定することで、X軸・Y軸に実際の半径の値を表示させることができます。
次に注目したいのはペアが密集している左下の領域、すなわち 0.0〜0.01 付近です。この部分を拡大して詳しく見てみましょう。
デフォルトでは、ヒストグラムを構築する際に、すべてのペアが収まるように自動的に表示範囲が設定されます。
この表示範囲を変更したい場合は、histogram メソッドの第1引数である x_range を指定することで、任意の範囲に調整することができます。
pd1.histogram((0, 0.01)).plot(colorbar={"type": "log"})
ヒストグラムの解像度を変更したい場合は、histogram メソッドの第2引数である x_bins を指定します。
デフォルトでは 128×128 の解像度でヒストグラムが描画されますが、ここではより詳細に表示するために、256×256 に設定してみましょう。
pd1.histogram((0, 0.01), 256).plot(colorbar={"type": "log"})
これらの図はMatplotlibの機能を使って描画されているため、matplotlib.pyplot.savefig を使って画像ファイルとして保存することができます。
たとえば、以下のように記述することで、図を pointcloud-pd1.png という名前で保存できます。
pd1.histogram((0, 0.01), 256).plot(colorbar={"type": "log"})
plt.savefig("pointcloud-pd1.png")
2次元のPDも同様に可視化しましょう.
pd2 = pdlist.dth_diagram(2)
pd2.histogram().plot(colorbar={"type": "log"})
この図では、death(Y軸)の値が 0.0125 を超えるあたりに、ペアが点在しているのが特徴的です。
数値データの取得¶
pd1 や pd2 が指すオブジェクトには births、deaths 属性があり、これらを参照することで birth time や death time を取得することができます。
pd1.births
array([0.00140906, 0.00139591, 0.00134116, ..., 0.01572219, 0.01709659,
0.01871588], shape=(3383,))
pd1.deaths
array([0.0015926 , 0.00164399, 0.00170775, ..., 0.0157662 , 0.01717465,
0.01880101], shape=(3383,))
この2つの差を計算するとlifetimeが得られます。
pd1.deaths - pd1.births
array([1.83534435e-04, 2.48078852e-04, 3.66588805e-04, ...,
4.40061409e-05, 7.80638251e-05, 8.51306753e-05], shape=(3383,))
lifetime のヒストグラムを表示してみましょう。
plt.hist(pd1.deaths - pd1.births, bins=100);
行末の ; は、plt.hist の返り値を表示せずに無視するためのテクニックです。
このヒストグラムから、多くの lifetime が非常に小さいことがわかります。 つまり、パーシステンス図の対角線付近に、多数のペアがノイズ的な情報として分布していることを示しています。
逆解析について¶
パーシステンス図に現れる各点は、元のポイントクラウドに存在するリング構造や空隙構造など、何らかの幾何的特徴に対応していると考えられます。
しかし、それが具体的にどのような構造なのかを特定するのは、実はそれほど簡単ではありません。このような解析手法は 逆解析(inverse analysis) と呼ばれます。
HomCloud には、以下のような逆解析機能が用意されています:
- Optimal volume
- Stable volume
- Optimal 1-cycle
- PH trees
それぞれに利点と欠点がありますが、最も汎用的に利用できるのは stable volume であり、本チュートリアルでもこれを使用します。
Stable volume は、optimal volume を発展させた手法で、小さなノイズによるリング構造の変化を考慮できる点が特徴です。
詳細については、以下の論文をご参照ください:
https://doi.org/10.1007/s41468-023-00119-8
まずは、パーシステンス図から1点を選び、それに対して逆解析を適用してみましょう。
ここでは、pd.nearest_pair_to メソッドを使って、点 $(0.0025, 0.008)$ に最も近い birth-death ペアを検索します。
pair = pd1.nearest_pair_to(0.0025, 0.008)
このオブジェクトは Pair クラスのインスタンスであり、birth time や death time などの情報を保持しています。
pair
Pair(0.0025623095880009357, 0.008135892057712316)
pair.lifetime()
np.float64(0.005573582469711381)
以下のように記述することで、stable volume を計算することができます。
ここで指定している 0.0002 は、想定されるノイズの大きさを表しています。
基本的には、データに含まれているノイズの性質を考慮して、このノイズスケールの値を設定する必要があります。
このパラメータの決定方法については、前述の論文で詳しく解説されています。
経験的には、lifetime や birth time の 1/10〜1/100 程度の値を指定すると、良好な結果が得られることが多いようです。
stable_volume = pair.stable_volume(0.00005)
boundary_points メソッドで、対応するリング構造に含まれる点の座標が得られます。
stable_volume.boundary_points()
[[0.24041004740553085, 0.3620263927982651, 0.3938519814000714], [0.017231099351335488, 0.10413818329710312, 0.6827502061744146], [0.07850470821281752, 0.31832538225761675, 0.2726051865809148], [0.1255383953826733, 0.0800900311964785, 0.7190957020829821], [0.1252375238953729, 0.20843145021582987, 0.37381081780757186], [0.27896903483481006, 0.2900038108788685, 0.3853792732744701], [0.11968719444459341, 0.46213675229879714, 0.6086047255929676], [0.10439142042970573, 0.12004955606294554, 0.3999294686167837], [0.07478379187666995, 0.1318144315592794, 0.6167927456566142], [0.241996106473225, 0.4429189262369284, 0.4639170406136188], [0.12423094383664512, 0.3622394365995112, 0.6448082172747804], [0.19443369660602383, 0.2643008696498902, 0.7685550494906768], [0.06800441912888222, 0.1305283961003515, 0.5058547294424975], [0.41898602615433533, 0.44362916692289767, 0.3710002303374377], [0.12195279248515212, 0.003239458152972219, 0.6061209669659768], [0.21608561791517278, 0.2918069725141198, 0.3219258262415402], [0.1894939883612946, 0.03095999913576697, 0.6762565272706147], [0.0708147888171643, 0.05566245889432686, 0.6142169362499716], [0.39171929078549805, 0.4405548898038244, 0.288435385498083], [0.006270880292196468, 0.10979896194917471, 0.5705032405071087], [0.3166872139448015, 0.38348372951946463, 0.33415602643779374], [0.3806980910779977, 0.4089441782551778, 0.43539259723846135], [0.15814254898557367, 0.44579878013749064, 0.4799628611505681], [0.17887993848973416, 0.42554668220768377, 0.6217943706156428], [0.09845847975583133, 0.26404248130005237, 0.7778474474038211], [0.3373945662185728, 0.46415106121781036, 0.4856613355037861], [0.08346167978435703, 0.26153343284444097, 0.3241366037151565], [0.03223409787023468, 0.08546385914763066, 0.4515319922985789], [0.03570453518233241, 0.17677934680678953, 0.6507549326106109], [0.08561651425017891, 0.42297356026275923, 0.528205350556188], [0.12886238414379725, 0.17512618876757002, 0.7438074952407614], [0.16660019888277078, 0.33716865816694763, 0.30111066188327684], [0.32035651808142795, 0.4751033316678963, 0.34029734006837487], [0.18678772331743787, 0.34044366488617717, 0.7226556446102448]]
len(stable_volume.boundary_points())
34
さらに、boundary メソッドを使用することで、どの点とどの点が接続されているかといった詳細な構造情報も取得できます。
これを3次元可視化してみましょう。
pl = pv.Plotter()
pl.add_mesh(stable_volume.to_pyvista_boundary_mesh())
pl.show()
<MagicMock name='mock()' id='134035003131328'>
リングが表示されます。

ポイントクラウドとリング構造を重ねて表示してみましょう。 この可視化には PyVista の機能を活用し、線の太さや色を調整することで、構造をより見やすく表現します。
pl = pv.Plotter()
pl.add_mesh(pv.PointSet(pointcloud))
pl.add_mesh(stable_volume.to_pyvista_boundary_mesh(), color="red", line_width=4)
pl.show()
<MagicMock name='mock()' id='134035003131328'>
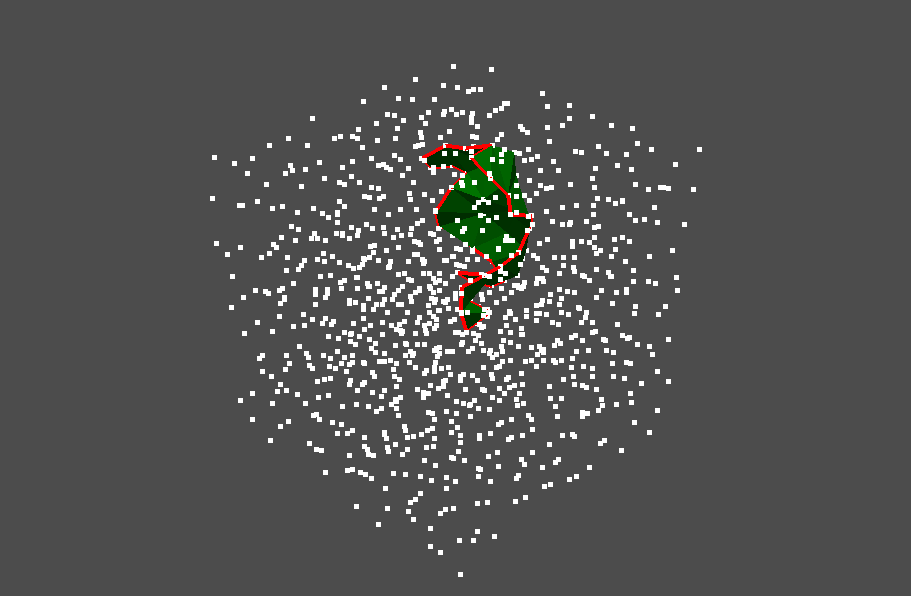
以下のように可視化されます。3次元データを回転させて見てみましょう。

stable volume の内部、つまりリング構造が囲んでいる領域の可視化も可能です。
pl = pv.Plotter()
pl.add_mesh(pv.PointSet(pointcloud))
pl.add_mesh(stable_volume.to_pyvista_boundary_mesh(), color="red", line_width=4)
pl.add_mesh(stable_volume.to_pyvista_volume_mesh(), color="green")
pl.show()
<MagicMock name='mock()' id='134035003131328'>
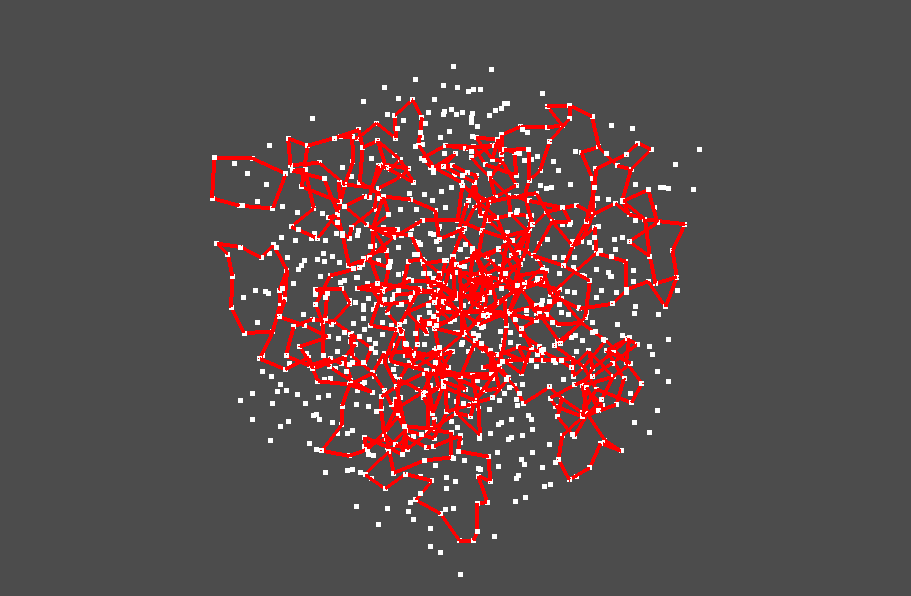
今度は、複数の点に対応する stable volume を同時に可視化してみましょう。
そのために、まずは birth < 0.004 かつ death > 0.006 を満たすようなペアを抽出します。 この条件に合致するペアは、以下のような方法で取り出すことができます。
pairs = [pair for pair in pd1.pairs() if pair.birth_time() < 0.004 and pair.death_time() > 0.006]
取り出したペアを数えます。
len(pairs)
54
では、実際に stable volume を計算し、可視化してみましょう。 今回は対象となるペアが 54個もあるため、計算にはそれなりの時間がかかります。
そこで、計算時間を短縮するために cutoff_radius を指定します。
これは、計算対象となる構造が存在すると予想される領域の大きさ(半径)をあらかじめ指定することで、不要な領域の計算を省略するための仕組みです。
この半径を正確に予測するのは簡単ではありませんが、今回のデータは全体の座標が 0〜1 の範囲に分布しているため、 その半分である 0.5 を目安として指定してみましょう。
%%time
stable_volumes = [pair.stable_volume(0.00005, cutoff_radius=0.5) for pair in pairs]
CPU times: user 10.3 s, sys: 127 ms, total: 10.5 s Wall time: 13.6 s
cutoff_radius=None とするとこの時間短縮機能が省略されるので,どのくらい計算時間が変わるか試してみるとよいでしょう。
次のセルで可視化します。
pl = pv.Plotter()
for stable_volume in stable_volumes:
pl.add_mesh(stable_volume.to_pyvista_boundary_mesh(),
color="red", line_width=4)
pl.add_mesh(pv.PointSet(pointcloud))
pl.show()
<MagicMock name='mock()' id='134035003131328'>
2次元パーシステンス図の逆解析について¶
次は、空洞(2次元ホモロジー) に対応する構造も可視化してみましょう。 ここでは、birth > 0.0125 かつ death − birth > 0.0005 を満たす領域に注目して解析を行います。
pairs = [pair for pair in pd2.pairs() if pair.birth_time() > 0.0125 and pair.lifetime() > 0.0005]
stable_volumes = [pair.stable_volume(0.000005) for pair in pairs]
pl = pv.Plotter()
for stable_volume in stable_volumes:
pl.add_mesh(stable_volume.to_pyvista_boundary_mesh(), color="green")
pl.add_mesh(pv.PointSet(pointcloud))
pl.show()
<MagicMock name='mock()' id='134035003131328'>
結構大きな空洞があるようです.

付録(発展的話題)¶
以下ではいくつか発展的なトピックについて紹介します.
頂点の命名¶
これまで boundary_points などで取得できる頂点情報は、XYZ座標のみでした。
多くの場合はこれで十分ですが、たとえば「4番目の点」や「12番目の炭素原子」といった情報で頂点を識別したい場面もあるでしょう。
そのような場合には、頂点に名前を付けることで、より柔軟な扱いが可能になります。
名前のルールはシンプルで、文字列であれば何でも使用可能です。
また、異なる頂点に同じ名前を付けることも可能です(HomCloud内部では名前が重複していても、個々の頂点を正しく識別できるため問題ありません)。
ここでは、1000個の頂点に "000" から "999" までの名前を順番に付けてみましょう。
names = [f"{n:03d}" for n in range(1000)]
from_alpha_filtion の vertex_symbols 引数にこの名前のリストを渡すことで、頂点に名前を付けることができます。
hc.PDList.from_alpha_filtration(pointcloud, vertex_symbols=names, save_boundary_map=True, save_to="pointcloud-with-names.pdgm")
PDList(path=pointcloud-with-names.pdgm)
それでは、先ほどと同様に stable volume を計算し、その頂点を確認してみましょう。boundary_points の代わりに boundary_points_symbols を使うと、ループ上の頂点の名前のリストを取得できます。
pd1 = hc.PDList("pointcloud-with-names.pdgm").dth_diagram(1)
pair = pd1.nearest_pair_to(0.0025, 0.008)
stable_volume = pair.stable_volume(0.0005)
stable_volume.boundary_points_symbols()
['392', '147', '154', '297', '170', '939', '442', '957', '832', '065', '580', '581', '582', '972', '080', '721', '480', '870', '872', '491', '236', '237', '750']
Optimal 1-cycle¶
Stable volume は、内部的に線形計画法を用いて計算されています。そのため、点の数が多い(たとえば 100,000 点以上)場合には、計算に時間がかかることがあります。
この問題に対応するため、HomCloud にはいくつかの高速化手法が用意されています。
1 次元のパーシステントホモロジー、つまりループ構造を解析する場合には、optimal 1-cycle という手法を使って、類似した情報をより高速に計算することが可能です。
ただし、この手法はある種の近似に基づいており、グラフ上の最短経路アルゴリズムによって計算されるため、パーシステントホモロジーの情報を完全に反映するものではありません。そのため、optimal volume や stable volume とは異なる結果が得られる可能性があります。
通常は stable volume の使用を推奨しますが、点の数が非常に多い場合には、optimal 1-cycle の利用を検討してみてください。
以下では、先ほどの例と同様に、点 (0.0025, 0.008) に最も近い点に対応する optimal 1-cycle を計算してみます。
この処理には optimal_1_cycle メソッドを使用します。stable volume におけるノイズ幅パラメーターに相当する値として、tolerance 引数を指定することができます。
pair = pd1.nearest_pair_to(0.0025, 0.008)
optimal_1_cycle = pair.optimal_1_cycle(torelance=0.0002)
可視化をします。stable volumeも一緒に計算してその違いを確認します。
stable_volume = pair.stable_volume(0.0002)
pl = pv.Plotter()
pl.add_mesh(optimal_1_cycle.to_pyvista_mesh(), color="red", line_width=4, opacity=0.7)
pl.add_mesh(stable_volume.to_pyvista_boundary_mesh(), color="green", line_width=4, opacity=0.7)
pl.add_mesh(pv.PointSet(pointcloud))
pl.show()
<MagicMock name='mock()' id='134035003131328'>
ループの一部は共通していますが、全体としては一致していないことがわかります。
次のようにして、optimal 1-cycleに含まれる辺の情報を得ることができます.
optimal_1_cycle.path()
[[[0.1255383953826733, 0.0800900311964785, 0.7190957020829821], [0.1894939883612946, 0.03095999913576697, 0.6762565272706147]], [[0.1255383953826733, 0.0800900311964785, 0.7190957020829821], [0.12886238414379725, 0.17512618876757002, 0.7438074952407614]], [[0.12886238414379725, 0.17512618876757002, 0.7438074952407614], [0.09845847975583133, 0.26404248130005237, 0.7778474474038211]], [[0.19443369660602383, 0.2643008696498902, 0.7685550494906768], [0.09845847975583133, 0.26404248130005237, 0.7778474474038211]], [[0.19443369660602383, 0.2643008696498902, 0.7685550494906768], [0.18678772331743787, 0.34044366488617717, 0.7226556446102448]], [[0.18678772331743787, 0.34044366488617717, 0.7226556446102448], [0.2422121988843836, 0.4155487622456445, 0.7075748545403898]], [[0.3058423940248698, 0.3873355905611001, 0.6845908848342269], [0.2422121988843836, 0.4155487622456445, 0.7075748545403898]], [[0.3058423940248698, 0.3873355905611001, 0.6845908848342269], [0.3756708152548601, 0.4044688174747033, 0.6376955289875823]], [[0.42889440234947107, 0.4185878889153811, 0.5784790134722813], [0.3756708152548601, 0.4044688174747033, 0.6376955289875823]], [[0.42889440234947107, 0.4185878889153811, 0.5784790134722813], [0.4598816637670946, 0.4162817757882945, 0.49121522961841546]], [[0.47167872450123804, 0.3563222306553614, 0.4586659808644902], [0.4598816637670946, 0.4162817757882945, 0.49121522961841546]], [[0.47167872450123804, 0.3563222306553614, 0.4586659808644902], [0.4643529555106557, 0.38891409104439234, 0.37758039548286215]], [[0.41872842706632685, 0.3117355628197983, 0.36357591323139904], [0.4643529555106557, 0.38891409104439234, 0.37758039548286215]], [[0.41872842706632685, 0.3117355628197983, 0.36357591323139904], [0.39907517145005755, 0.2831943114569727, 0.4368217260059085]], [[0.34941504868714357, 0.20455144955416626, 0.4210171084082195], [0.39907517145005755, 0.2831943114569727, 0.4368217260059085]], [[0.34941504868714357, 0.20455144955416626, 0.4210171084082195], [0.296676557056482, 0.1310208804226647, 0.43046852751189457]], [[0.296676557056482, 0.1310208804226647, 0.43046852751189457], [0.2927591672470917, 0.11598883729155451, 0.5038453999131621]], [[0.21062788160417378, 0.11633529728096403, 0.5162505592185838], [0.2927591672470917, 0.11598883729155451, 0.5038453999131621]], [[0.21062788160417378, 0.11633529728096403, 0.5162505592185838], [0.1563951908464587, 0.13629542421604657, 0.594303189697284]], [[0.1563951908464587, 0.13629542421604657, 0.594303189697284], [0.07478379187666995, 0.1318144315592794, 0.6167927456566142]], [[0.0708147888171643, 0.05566245889432686, 0.6142169362499716], [0.07478379187666995, 0.1318144315592794, 0.6167927456566142]], [[0.0708147888171643, 0.05566245889432686, 0.6142169362499716], [0.12195279248515212, 0.003239458152972219, 0.6061209669659768]], [[0.1894939883612946, 0.03095999913576697, 0.6762565272706147], [0.12195279248515212, 0.003239458152972219, 0.6061209669659768]]]
PHTrees¶
2 次元のパーシステントホモロジー、つまり空隙構造を解析する場合には、PHTrees(Persistence Trees) を利用することができます。 この手法は、数学的な双対性(duality)を活用することで、2 次元のパーシステンス図を逆解析込みで高速に計算することを可能にします。
PHTrees を計算してみましょう。
save_phtrees=True という引数を指定することで、パーシステンス図の計算と同時に PHTrees の計算も行われます。
hc.PDList.from_alpha_filtration(pointcloud,
save_to="pointcloud-phtrees.pdgm",
save_phtrees=True,
save_boundary_map=True)
PDList(path=pointcloud-phtrees.pdgm)
次のようにして PHTrees を読み込みます。
phtrees = hc.PDList("pointcloud-phtrees.pdgm").dth_diagram(2).load_phtrees()
先ほどの例と同様に、birth > 0.0125 かつ death - birth > 0.0005 を満たすペアに注目してみましょう。
PHTreesは内部的には木構造(厳密には複数の根を持つので森構造です)をしていて、各ペアと木構造のノードが対応しています.
stable_volumes = [node.stable_volume(0.000005) for node in phtrees.all_nodes if node.birth_time() > 0.0125 and node.lifetime() > 0.0005]
可視化します。
pl = pv.Plotter()
for sv in stable_volumes:
pl.add_mesh(sv.to_pyvista_mesh(), color="green")
pl.add_mesh(pv.PointSet(pointcloud))
pl.show()
<MagicMock name='mock()' id='134035003131328'>
形の情報は boundary_points メソッドや boundary メソッドなどで取り出せます.
stable_volumes[0].boundary_points()
[[0.6994453732085671, 0.13144153221118793, 0.5796928995428492], [0.601928672387627, 0.08671998624231247, 0.6583244074235682], [0.6994366874462885, 0.002942826575932589, 0.8357930347813906], [0.7794027918486173, 0.20026894822385644, 0.7191885213983096], [0.7064811805219977, 0.0026801020745949033, 0.5743802347904137], [0.8393149713230158, 0.04786549991547495, 0.6049694308550129], [0.8709165597839585, 0.12038179501110158, 0.6840309300141862]]
その他にも、PHTrees の木構造を活用したさまざまな解析が可能です。
詳しくは、HomCloud の API マニュアルの PHTrees の項をご参照ください。
初期半径¶
粒子に固有の大きさがあるようなデータ、たとえば複数種類の原子を含む原子配置データを解析したい場合には、それぞれの半径を反映した解析が可能です。
$i$ 番目の原子が半径 $r_i$ を持つとき、配列 $w = [r_0^2, \ldots, r_{N-1}^2]$ を用意することで、HomCloud においてその情報を解析に組み込むことができます。
※HomCloud の仕様では、半径は 2乗 して指定する必要があります。
以下の例では、1000 個の粒子のうち、最初の 500 個の初期半径を 0.001、残りの 500 個の半径を 0.005 としています。
weights = np.concatenate([np.full(500, 0.001), np.full(500, 0.005)])
hc.PDList.from_alpha_filtration(pointcloud, weight=weights,
save_boundary_map=True, save_to="pointcloud-weights.pdgm")
PDList(path=pointcloud-weights.pdgm)
pd1 = hc.PDList("pointcloud-weights.pdgm").dth_diagram(1)
pd1.histogram().plot(colorbar={"type": "log"})
初期半径を正の値に設定すると、birth や death の値が負になる場合があります。これは、解析において「球を初期半径から縮めていく」状況を想定しているためです。
HomCloud では、各粒子の半径は次の式で定義されます:
$$ \text{半径} = \sqrt{\alpha + r_i^2} $$
ここで、$\alpha$ はフィルトレーションパラメータであり、これを変化させることで球の大きさが変化します。初期状態では $\alpha = 0$ であり、そこから $\alpha$ を小さくすることで球を縮める(=負のスケールでの構造を観察する)ことができます。
BackgroundPlotter¶
pyvista と Jupyter Notebook を組み合わせた可視化は便利ですが、場合によってはパフォーマンスが不足することがあります。
特に、大規模なボクセルデータを可視化する際には、表示までに時間がかかることがあります。これは、おそらくバックエンド(Python)とフロントエンド(ブラウザ + JavaScript)の間の通信に時間がかかっているためと考えられます。
また、Jupyter Notebook に埋め込まれた表示が使いにくいと感じることもあるでしょう。
そのような場合には、pyvistaqt の BackgroundPlotter を使うのがおすすめです。
pyvistaqt を利用するには、pyqt6 をインストールしておく必要があります。
なお、BackgroundPlotter は通常の pyvista の Plotter とは共存できないため、どちらか一方のみを使用するようにしてください。
import pyvistaqt as pvqt
pl = pvqt.BackgroundPlotter()
pl.add_mesh(pv.PointSet(pointcloud))
pl.show()
<MagicMock name='mock().show()' id='134034304441264'>